http://pic.mbu.iisc.ernet.in/
蛋白质相互作用计算器(PIC)是可识别各种相互作用的服务器。 例如蛋白质内或复合物中蛋白质之间的二硫键,疏水相互作用,离子相互作用,氢键,芳族-芳族相互作用,芳族-硫相互作用和阳离子-阳离子相互作用。 它还确定可及的表面积以及残基与蛋白质表面的距离。 输入应为蛋白质数据库(.pdb)格式。 互动是根据经验或半经验规则集计算的。
疏水作用力分析
http://curie.utmb.edu/prosurf.html
InterProSurf ,
使用“ PDB Complex”选项查找蛋白质数据库中存放的蛋白质复合物结构中的界面残基使用
使用“User Complex”选项查找您感兴趣的蛋白质复合物中的界面残基
使用补丁分析和聚类预测单体蛋白质表面上的相互作用残基分析
https://www.ebi.ac.uk/pdbe/pisa/
PDBePISA是用于探索高分子界面的交互式工具。
使用PDBePISA,您可以:检索整个PDB存档的预先计算的结果。
以交互方式计算作为PDB或mmCIF文件上传的结构的结果这些计算的结果包括:大分子表面和界面的可能的四级结构(装配体)的结构和化学性质,其结构和化学性质以及可能的解离模式在PDB档案库中搜索由以下分子形成的特定界面 结构同系物。
使用多种选择搜索PISA数据库中的预计算结果,例如:多聚态,对称数,空间群,可及/埋藏的表面积,离解自由能,盐桥和二硫键的存在与否,同构 类型,配体关键字使用Rasmol(Unix / Linux平台),Rastop(MS Windows机器)和Jmol(与平台无关的服务器端Java查看器)评估大分子界面的重要性(生物学作用)并可视化结构,界面和装配体
。
Pymol分析


 旧版首页
旧版首页 全新首页
全新首页
 NCS
NCS  学术网站链接
学术网站链接  蛋白质化学工具
蛋白质化学工具  蛋白质平台
蛋白质平台  实用工具网站
实用工具网站 
 学术动态
学术动态  人文建设
人文建设  学术交流
学术交流  人物随感
人物随感  其他新闻
其他新闻  学术周边
学术周边  科学大事件
科学大事件 
 Online PDB
Online PDB  2024年
2024年  2023年
2023年  2022年
2022年  2021年
2021年  2020年
2020年  2019年
2019年  2018年
2018年  2017年
2017年  2016年
2016年  2015年
2015年  2014年
2014年  Prior to 2014
Prior to 2014 
 实验室概况
实验室概况  研究方向
研究方向 
 Thesis Defence
Thesis Defence  Lab Meeting
Lab Meeting  Fun Time
Fun Time  Awards
Awards  Talk
Talk  Notice
Notice  Experiment notebook
Experiment notebook  实验室安全教育
实验室安全教育 
 Groupleader
Groupleader  Associate Professor
Associate Professor  Postdoctoral Fellows
Postdoctoral Fellows  Ph.D Candidates
Ph.D Candidates  Master Candidates
Master Candidates  Undergraduate
Undergraduate  Collaborator
Collaborator  Former Lab Members
Former Lab Members 
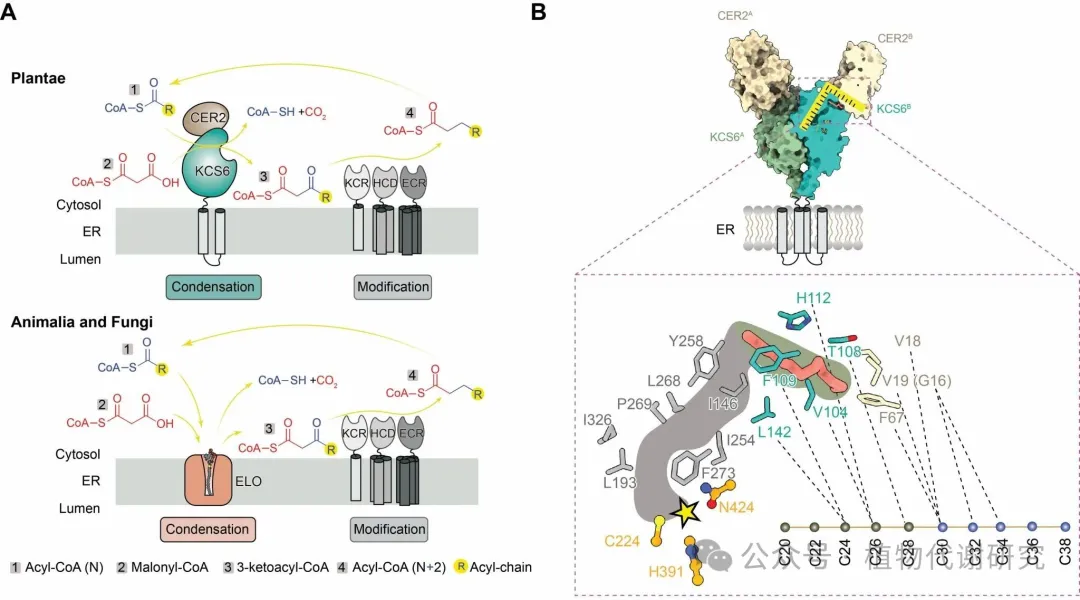
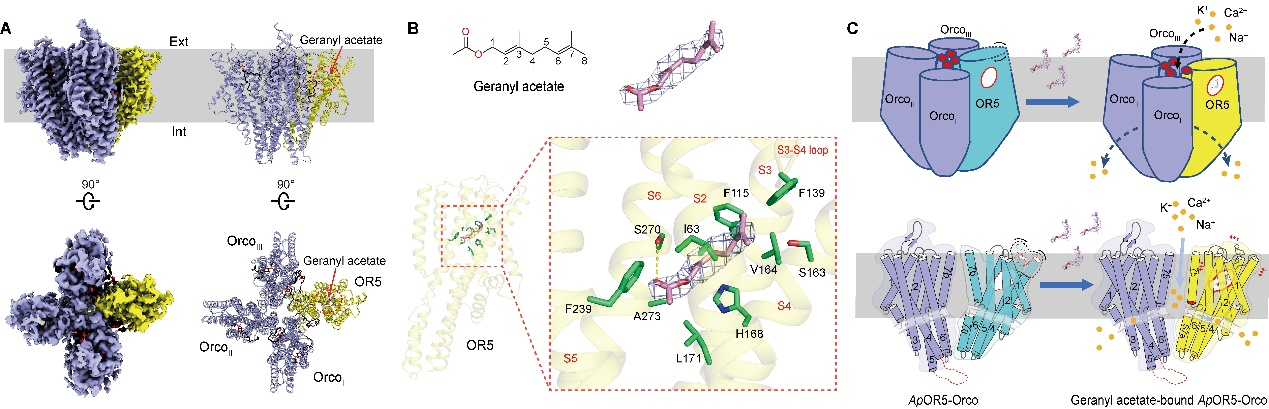
 鄂公网安备 42011102000808号
鄂公网安备 42011102000808号 
