南湖新闻网讯(通讯员 洪思行)3月17日,Nature Communications在线发表了我校作物遗传改良国家重点实验室、生命科学技术学院结构生物学研究团队解析叶绿体DNA重组修复的最新研究成果。成果论文以“Structural insights into sequence-dependent Holliday junction resolution by the chloroplast resolvase MOC1”为题,解析了植物叶绿体Holliday junction解离酶序列特异性识别和切割DNA的分子机制。
Holliday junction(HJ)是由英国生物学家Robin Holliday提出的DNA分子间的十字交叉结构,是同源重组过程中一类重要的DNA中间体,其对于DNA损伤修复以及保持物种遗传多样性等具有重要意义。而这种特殊的DNA结构必须在解离酶的作用下及时解离成双链线性的DNA才能保持基因组DNA的稳定性。HJ解离酶在噬菌体、细菌、真菌、植物和动物中是广泛存在的,虽然这类酶的序列同源性不高,但它们发挥作用的机理有一定的相似性。然而,HJ解离酶对DNA序列依赖的结构解离的分子机制却一直未知。
在本研究中,研究者解析了植物叶绿体HJ解离酶MOC1单体以及结合HJ的复合体的晶体结构。MOC1以二聚体形式发挥功能。其中,HJ镶嵌在二聚体MOC1的两个凹槽中。通过结构分析,鉴定了其中四个酸性氨基酸组成了催化四连体,负责切割DNA碱基间的磷酸二酯键。其中最重要的发现是,本研究鉴定了一个重要的DNA碱基识别环(base recognition loop, BR loop),其中有两个关键的氨基酸参与了junction位置C-G碱基对的打开,以及特定的DNA碱基识别,从而决定了序列识别以及切割的特异性。综合晶体结构解析和系统的生化实验验证,本研究从分子水平上对于HJ解离酶序列特异性切割HJ的科学问题进行了解答。
通过结构序列比对分析,研究者发现这类BR loop在其他物种的HJ解离酶,如细菌RuvC和酵母Ydc2中是保守存在的。因此,本研究鉴定的BR loop对其它类型特异性识别和切割DNA的HJ解离酶,具有普通的参考意义。
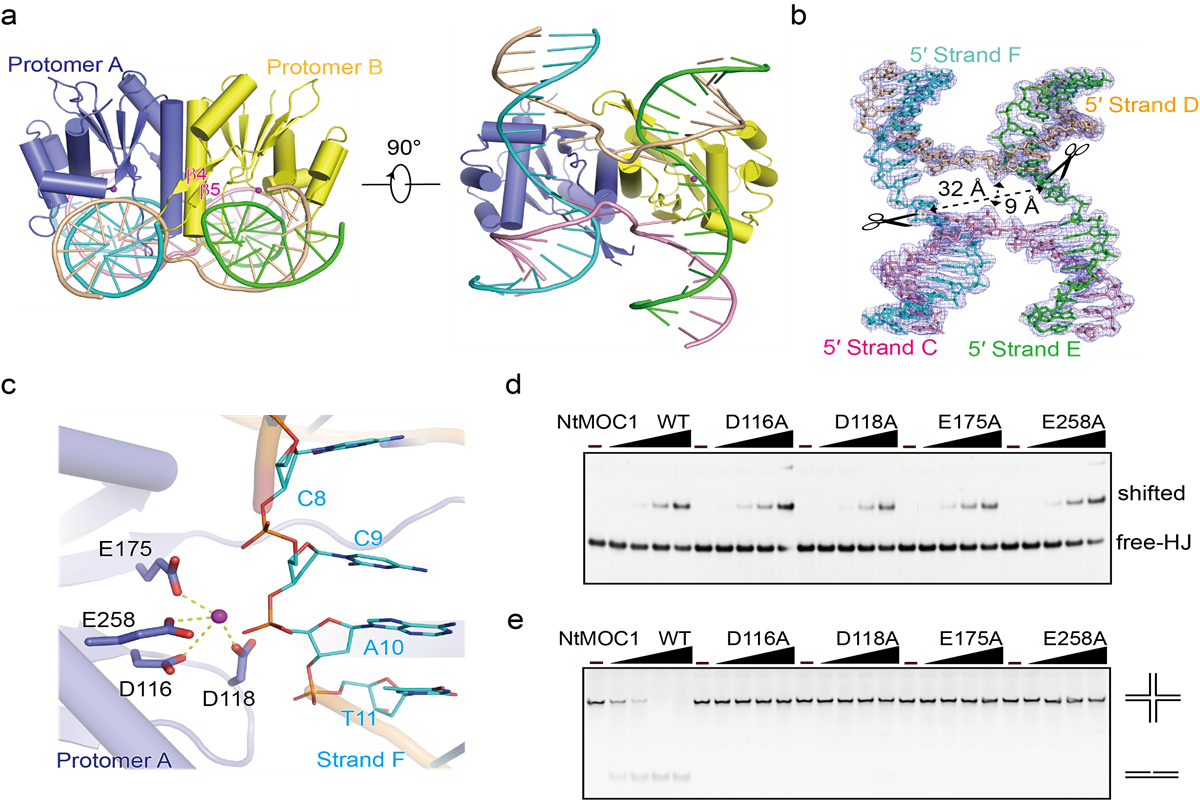
叶绿体MOC1-HJ复合物的晶体结构以及催化机制分析
博士闫俊杰、研究生洪思行为本论文共同第一作者,殷平教授为论文通讯作者。校级蛋白质平台为该研究的开展提供了有力支持。上海同步辐射光源(SSRF)为数据收集提供了帮助。该研究受到了科技部基金、国家自然科学基金-优青基金和青年基金、华中农业大学科技自主创新基金、中国博士后基金的共同资助。
据悉,本研究工作是该研究团队在叶绿体基因表达调控领域系列研究工作的新突破。团队揭示了叶绿体转录后调控蛋白PPR家族特异性识别RNA碱基的分子机制及潜在应用(Shen et al., 2015, Molecular Plant; Shen et al., 2016, Nature Communications; Yan et al., 2019, Nucleic Acids Research);揭示了叶绿体RNA编辑因子MORF蛋白调控靶标RNA识别的分子机制(Yan et al., 2017, Nature Plants;Yan et al., 2018,Science China Life Sciences)
审核人:殷平


 旧版首页
旧版首页 全新首页
全新首页
 NCS
NCS  学术网站链接
学术网站链接  蛋白质化学工具
蛋白质化学工具  蛋白质平台
蛋白质平台  实用工具网站
实用工具网站 
 学术动态
学术动态  人文建设
人文建设  学术交流
学术交流  人物随感
人物随感  其他新闻
其他新闻  学术周边
学术周边  科学大事件
科学大事件 
 Online PDB
Online PDB  2024年
2024年  2023年
2023年  2022年
2022年  2021年
2021年  2020年
2020年  2019年
2019年  2018年
2018年  2017年
2017年  2016年
2016年  2015年
2015年  2014年
2014年  Prior to 2014
Prior to 2014 
 实验室概况
实验室概况  研究方向
研究方向 
 Thesis Defence
Thesis Defence  Lab Meeting
Lab Meeting  Fun Time
Fun Time  Awards
Awards  Talk
Talk  Notice
Notice  Experiment notebook
Experiment notebook  实验室安全教育
实验室安全教育 
 Groupleader
Groupleader  Associate Professor
Associate Professor  Postdoctoral Fellows
Postdoctoral Fellows  Ph.D Candidates
Ph.D Candidates  Master Candidates
Master Candidates  Undergraduate
Undergraduate  Collaborator
Collaborator  Former Lab Members
Former Lab Members 

 鄂公网安备 42011102000808号
鄂公网安备 42011102000808号 
